製品の特長 - ランダムインテグレーション解析「RAISING」
対象遺伝子のホストゲノム上の挿入位置の決定手法
国立感染症研究所とファスマックで共同り開発した外来DNAの挿入位置を決定する方法です。
外来DNAが目的位置も含め、ランダムにホストゲノムへ挿入(ランダムインテグレーション)された位置を決定することが可能です。
ウイルスのクロナリティ解析、ウイルスベクター挿入部位、創薬ターゲットとなりうる融合遺伝子の検索、ゲノム編集におけるオフターゲットノックインなど、用途は多岐に渡ります。 - ランダムインテグレーション解析「RAISING」
ファスマック社のNGS解析担当者宛に解析条件(対象生物、外来DNA情報、サンプルの発送状態、解析目的)をメールに記載して頂きご連絡ください。
E-mail:ngs@fasmac.co.jp
その後、お見積書をお送りさせて頂きますので、内容がよろしければ専用のオーダーシートに必要事項をご記入の上、見積番号とともにご注文ください。
仕様 - ランダムインテグレーション解析「RAISING」
| サンプル種類 | 精製済みのゲノムDNA(RNA処理済み) |
|---|
| 解析法 | Miseq |
|---|
| 報告内容 | 解析データをユーザー様にご提供 |
|---|
| 目安納期 | お問い合わせ |
|---|
| その他 | ①サンガーシーケンスプラン: ¥120,000(8検体あたり)
②NGSプラン:
8検体あたり: ¥250,000
リファレンスマッピングによる挿入位置の決定: ¥20,000(/ターゲット)
アノテーション情報の取得: ¥20,000(/ターゲット)
|
|---|
| メーカー希望小売価格(税別) | お問い合わせ下さい |
|---|
表示価格は代表的な仕様のものです。詳細はお問い合わせください。
※リンク先で情報を閲覧する際に、ログイン・フォーム入力等を求められる可能性がございます。
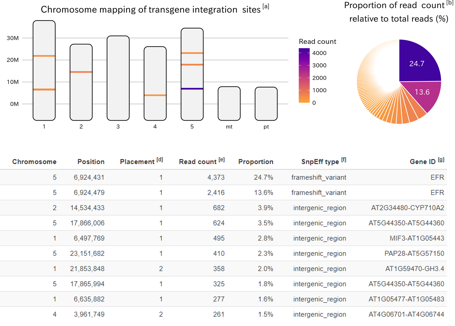
NGSによる挿入位置の特定、遺伝子アノテーションの結果。リード情報とあわせてランキングテーブルを作成いたします。
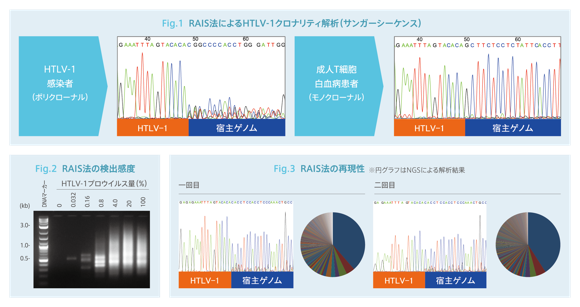
HTLV-1(ヒトT細胞白血病ウイルス)クロナリティ解析に応用した例。クロナリティの度合いを定量化するための解析ソフト「CLOVA」を利用することで、 サンガーシーケンス結果からクロナリティの度合いを定量化できます。

CRISPR/Casにより作製したノックインマウスについて、RAISING法およびNGSの組み合わせによりノックイン効率およびオフターゲットサイトの検出を試みました。
ノックインに用いたドナーDNAがオンターゲットに加えて、別の位置へも挿入されていることが示唆されました。